皮脂RNAにパーキンソン病患者に特有の情報が含まれることを発見
皮脂RNA情報と機械学習モデルによる新たな検査方法の可能性
2021.09.21
順天堂大学大学院医学研究科神経学の斉木臣二先任准教授、服部信孝教授、花王株式会社生物科学研究所、株式会社Preferred Networks(以下、PFN)らの研究グループは、パーキンソン病*1患者皮脂中のRNA(リボ核酸)*2に病態と関連した特有の情報が含まれることを発見しました。さらに皮脂RNA情報を用いた機械学習モデル*3がパーキンソン病の診断方法になりうることを明らかにしました。これらの結果は、皮脂RNAという簡便かつ侵襲を伴わずに採取可能な生体成分を用いたパーキンソン病の新たな検査方法の可能性を示した画期的な成果で、病態に基づく早期診断につながる点に意義があります。本成果は、英国科学雑誌「Scientific Reports」のオンライン版で公開されました。
本研究成果のポイント
- パーキンソン病患者には特有の皮脂RNA情報が存在
- パーキンソン病患者ではミトコンドリアの機能と関連した皮脂RNAが変化
- 皮脂RNA情報を用いた機械学習モデルがパーキンソン病早期診断法に
背景
パーキンソン病は有病率が10万人あたり約140人に上る日本で2番目に多い神経変性疾患で、運動に関する症状や自律神経障害、認知機能低下が徐々に進行します。現在のところパーキンソン病を根治するための治療方法は存在していませんが、早期に確定診断を行い、適切な治療を継続することで症状をコントロールすることができます。しかし、パーキンソン病の診断には専門的かつ複雑な検査が必要であるため、より簡便な検査方法が求められています。
パーキンソン病では皮脂の増加を伴う脂漏性皮膚炎などいくつかの皮膚症状が高頻度に併発することが知られています。そこで今回、「皮脂にはパーキンソン病と関連した情報が含まれる」との仮説を立て、パーキンソン病患者の病状を正確に反映するバイオマーカーを探索する順天堂大学の研究グループ、皮脂RNA*4の網羅的解析技術を保有する花王、機械学習や深層学習などの人工知能関連技術を保有するPFNは3者で共同研究を実施しました。
内容
研究グループは、軽症パーキンソン病患者*5を対象として2回の独立した試験を設定し、グループ1(未治療のパーキンソン病患者7名、健常者13名)、グループ2(未治療および内服加療中のパーキンソン病患者46名、健常者50名)の皮脂RNA情報の比較を行いました。皮脂RNAは1枚のあぶらとりフィルムを用いて顔全体から採取した皮脂から抽出することができます。抽出された皮脂RNAを用いて次世代シーケンサーによるRNA発現量を網羅的に解析し、含まれる情報の抽出や機械学習モデルの構築を行いました (図1)。
 図1: 皮脂RNA情報の取得と機械学習モデルの構築方法
図1: 皮脂RNA情報の取得と機械学習モデルの構築方法
あぶらとりフィルム1枚を用いて顔全体から皮脂を採取します。採取した皮脂からRNAを抽出し、次世代シーケンサーを用いて網羅的にRNA発現量を測定します。決定されたRNA発現量をパーキンソン病患者と健常者の間で比較することで、どのような身体や細胞の状態が反映されているかなどの情報の取得や、機械学習モデルの構築などを行います。
皮脂RNA解析の結果、グループ1・グループ2それぞれにおいて約4000種のRNAの情報が得られました。パーキンソン病患者において大きく変化していた約200~400種のRNAに注目したところ、パーキンソン病の病態と密接に関係するミトコンドリア*6に関連した複数のRNAが増加する傾向が示されました (図2)。このことから、パーキンソン病患者の皮脂RNAには健常者とは異なる情報が含まれること、さらにそれら皮脂RNAから得られた情報が既に知られているパーキンソン病の病態に関連した変化と矛盾していないことが示されました。
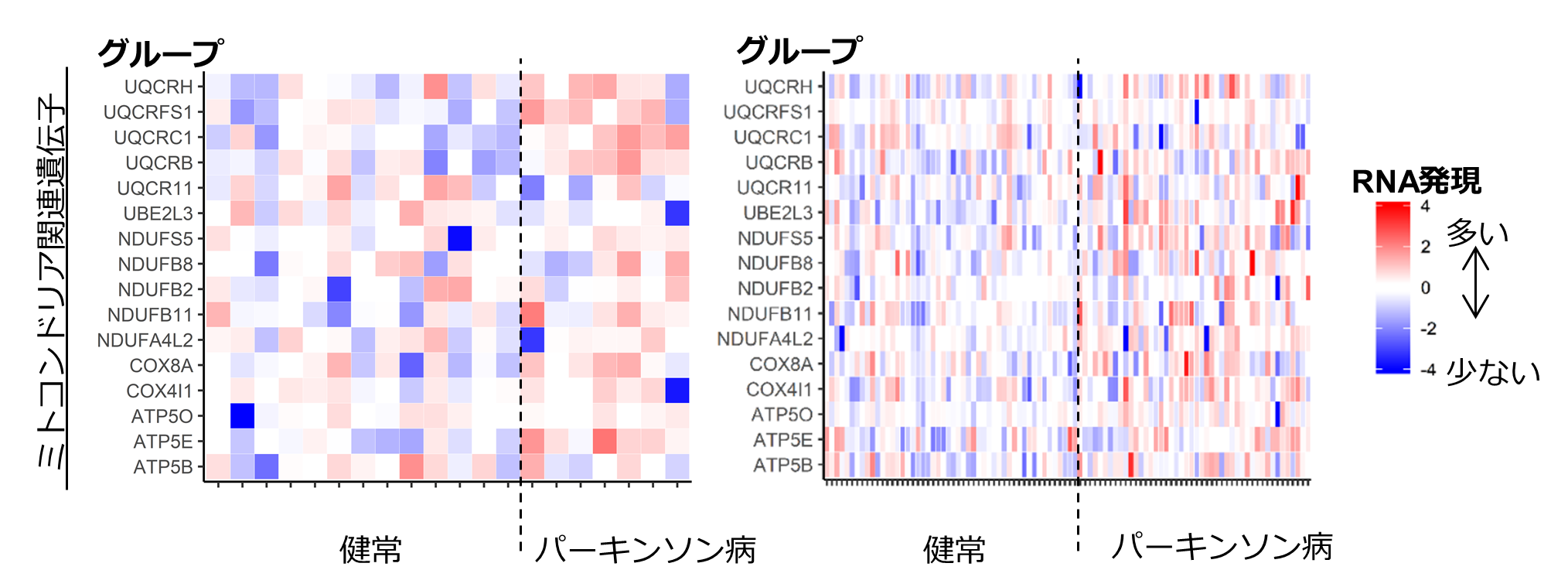 図2: ミトコンドリア機能に関連する皮脂RNA発現量の変化
図2: ミトコンドリア機能に関連する皮脂RNA発現量の変化
グループ1(パーキンソン病患者7名、健常者13名)、グループ2(パーキンソン病患者46名、健常者50名)のミトコンドリア機能に関連する皮脂RNAの変化値を示しています。中間のRNA発現量を白で示し、RNAが多いほど赤く、少ないほど青くなるように示しています。
次に、皮脂RNAの情報とExtremely Randomized Trees*7と呼ばれる機械学習モデルによってパーキンソン病を判別できるか検証しました。グループ1・2を統合して解析を行なった結果、皮脂RNA・年齢・性別情報を用いてパーキンソン病を判別することが可能であることが示されました(図3、橙色線)。また、同じ方法を用いてパーキンソン病の重症度*5を予測し、その予測された重症度の数値と皮脂RNA・年齢・性別情報を組み合わせて機械学習モデルを構築することによって、より精度よくパーキンソン病を判別することが可能でした(図3、青色線)。これらの結果から、皮脂RNAに含まれる情報を用いて機械学習モデルを構築することで、パーキンソン病を精度よく判定することができることが示されました。
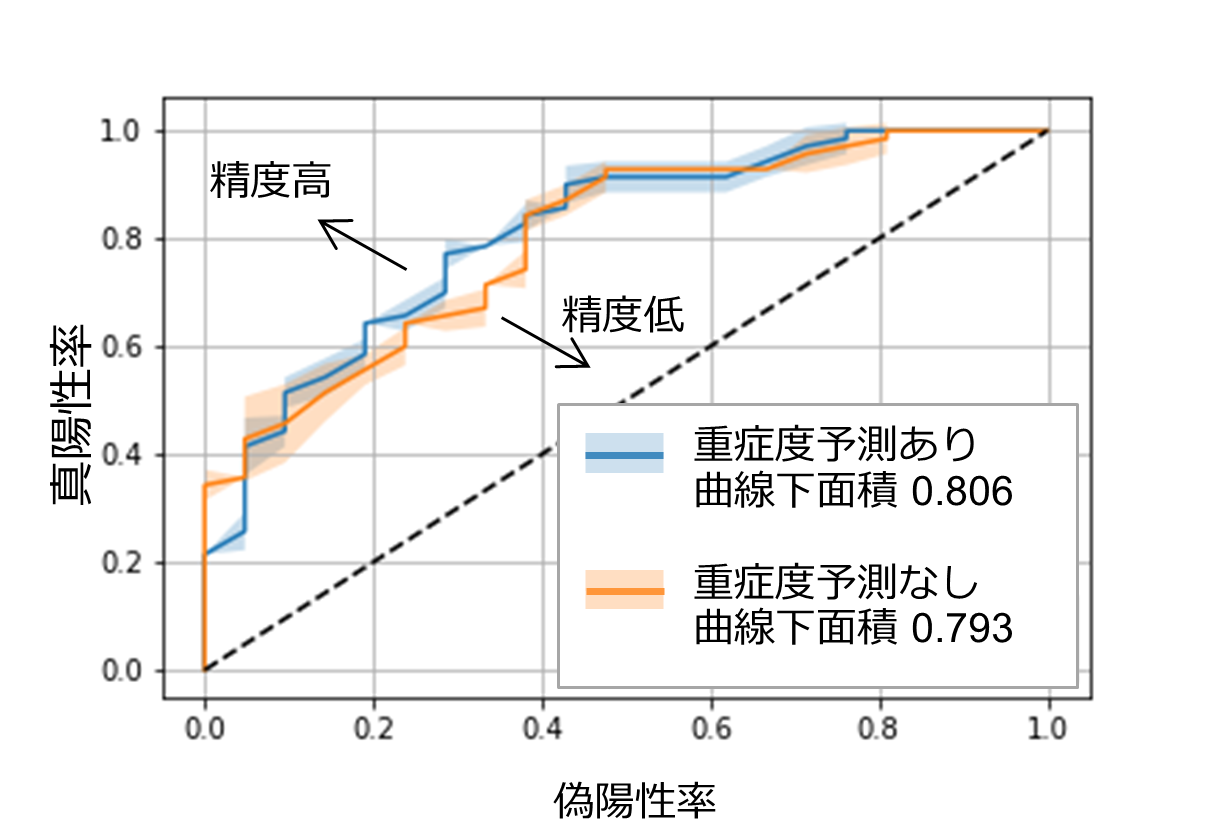 図3: 皮脂RNA情報と機械学習を用いたパーキンソン病の判別
図3: 皮脂RNA情報と機械学習を用いたパーキンソン病の判別
パーキンソン病患者と健常者の判別結果をReceiver Operating Characteristic (ROC)曲線*8で示しています。グラフ上の線が左上に近づくほど判別性能が高く、点線で示す対角線に近づくほど判別性能が低くなります。判別性能を表す曲線下面積は皮脂RNA、年齢、性別情報を用いた場合に0.793となり、これらに重症度予測結果を加えることで0.806とさらに高い性能が得られました。
今後の展開
本成果から皮脂RNA情報と機械学習モデルを組み合わせることにより、パーキンソン病の早期診断が可能となることが期待されます。この方法では、試料として用いる皮脂RNAをあぶらとりフィルム1枚を用いて侵襲を伴うことなく誰でも採取することが可能であることから、簡便なパーキンソン病の検査方法が提供できるようになれば、早期診断や先制医療開発の一助となるものと考えられます。
パーキンソン病の診断は、鑑別しなければならない類似の疾患が存すること、皮脂RNAの変化に関しても日々の生活などの外的要因が完全には精査されていないといった課題があります。研究グループでは、類似の疾患との鑑別診断が可能な機械学習モデルの構築や、精度向上のために制御が必要な日常生活の影響について検討を続けており、パーキンソン病の新たな検査方法の開発を目指しています。
用語解説
*1 パーキンソン病
進行性の中脳黒質神経細胞脱落を特徴とする神経変性疾患で、わが国の患者数は14万人とされる。高齢になるほど発症率が高まるため、2030年には全世界で1400万人が罹患すると予測されている。
*2 RNA (リボ核酸)
RNAはDNAの情報に基づき、生体内で合成される分子である。DNAに含まれる情報は基本的に一生不変だが、RNA情報は生体内の状態や外部の環境に応じて絶えず変化することから、動的な生体情報を得ることが可能な分子とされる。酵素や筋肉など生体内で様々さまざまな働きをするタンパク質はこのRNAの情報に基づいて合成されている。
*3 機械学習
機械学習は大量のデータを統計学的に処理することで、データに含まれるパターンやルールを見出すための手法である。一般に人工知能とよばれる技術の一つに機械学習があり、機械学習の手法の一つに近年注目されているディープラーニングなどがある。
*4 皮脂RNA
皮脂に含まれるRNAを指す。皮脂は皮脂腺に存在する脂腺細胞から全分泌と呼ばれる特殊な機構で分泌されている。脂腺細胞は全分泌において細胞内のすべての成分を放出するとされており、分泌された皮脂の中には脂質だけではなく細胞成分の一つであるRNAも含まれることが報告されている。さらに、皮脂はRNA分解酵素 (RNase)の活性を阻害する作用を持つとされ、分解を免れたRNAが解析可能な品質のまま皮脂に保持されているとされる。
*5 パーキンソン病の重症度
パーキンソン病の重症度の指標として「ヘーン-ヤールの重症度(I~V度)分類」が用いられる。I度が最も軽症であり、V度が最も重症である。I度およびII度をまとめて軽症として扱う場合がある。
*6 ミトコンドリア
ミトコンドリアは細胞のエネルギー産生に関与する細胞内小器官である。パーキンソン病患者の脳ではミトコンドリアのタンパク質の機能不全によって神経細胞死が起こるとされている。
*7 Extremely Randomized Trees
Extremely Randomized Treesは機械学習の手法の一つである。機械学習の中でも頻繁に用いられるRandom Forestと呼ばれる手法から派生したアルゴリズムであり、 Random Forestよりも効率的にモデルを構築できるという利点を持つとされる。
*8 Receiver Operating Characteristic (ROC)曲線
縦軸を真陽性率、横軸を偽陽性率として、カットオフ値を変化させながらプロットした曲線図。ある検査法がどれくらい有用であるかという性能を評価するために用いられる手法であり、0.5-1.0の間の値をとる曲線下面積が1.0に近いほど診断性能が高いと判断される。
原著論文
本研究成果は、英国科学雑誌の「Scientific Reports」誌のオンライン版に9月20日付で公開されました。
英文タイトル: Non-invasive diagnostic tool for Parkinson’s disease by sebum RNA profile with machine learning
日本語訳: 皮脂RNA情報と機械学習を用いたパーキンソン病の非侵襲的な診断方法
著者: Yuya Uehara1,2,5, Shin-Ichi Ueno3,5, Haruka Amano-Takeshige3,5, Shuji Suzuki4, Yoko Imamichi3, Motoki Fujimaki3, Noriyasu Ota1, Takatoshi Murase1, Takayoshi Inoue1, Shinji Saiki3, Nobutaka Hattori3 (5: Joint 1st authors)
著者(日本語表記): 上原裕也1,2,5、上野真一3,5、竹重遥香3,5 、鈴木脩司4、今道洋子3、藤巻基紀3、太田宣康1、村瀬孝利1、井上高良1、斉木臣二3、服部信孝3 (5: 共同筆頭著者)
所属: 1花王株式会社生物科学研究所、 2順天堂大学大学院医学研究科皮膚科学・アレルギー学、3順天堂大学大学院医学研究科神経学、 4株式会社Preferred Networks
DOI: 10.1038/s41598-021-98423-9
本研究は、JSPS科研費(研究代表者:斉木臣二: JP15H04843, JP18H02744, JP18KT0027, JP18KK0242)と共に、国立研究開発法人日本医療研究開発機構(AMED)の革新的先端研究開発支援事業(AMED-CREST)「疾患における代謝産物の解析および代謝制御に基づく革新的医療基盤技術の創出」研究開発領域*(研究開発総括:清水孝雄)における研究開発課題「パーキンソン病の代謝産物バイオマーカー創出およびその分子標的機構に基づく創薬シーズ同定」(研究開発代表者:服部信孝)による支援を受けて行われました。
また、本研究に協力頂きました患者さんのご厚意に深謝いたします。
*本研究開発領域は、平成27年4月の日本医療研究開発機構の発足に伴い、国立研究開発法人科学技術振興機構(JST)より移管されたものです。